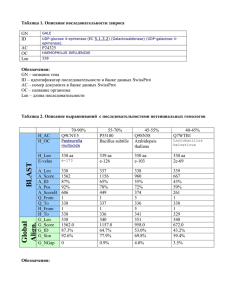
Сравнение множественного выравнивания, полученного с помощью программы
реклама

Сравнение множественного выравнивания, полученного с помощью программы ClustalW, с «биологически правильным» выравниванием из BaliBase. Выравнивание, с которым проводилось сравнение - 1pii. Сравниваемые последовательности (ID): TRPC_ACICA, TRPC_PHYPR, TRPC_HALVO, TRPC_METTH. Примечание, последовательности для выравнивания с помощью ClastalW брались из банка данных SwissProt по Accession Number последовательностей из 1pii документа. Оказалось, что по AC для TRPC_ACICA в банке SwissProt нашлась последовательность не TRPC_ACICA, а TRPC_ACIAD. Последовательности же с ID TRPC_ACICA там не обнаружилось. Проведя выравнивание последовательности TRPC_ACICA из документа 1pii с последовательностью TRPC_ACIAD из SwissProt, я обнаружил, что они отличаются только одной делецией/вставкой в начале (Identity 97%). Поэтому я условно считаю их одной и той же последовательностью. По данным из Balibase минимальный процент совпадения последовательностей 31, максимальный – 35. Сравнение проводилось по 151-175 колонкам (фрагмент длинной 25 колонок). Указанный участок выравнивания из BaliBase TRPC_ACICA : VACLsdqQLEEMSKTAFEYDLDVLV TRPC_PHYPR : VTLLskeQLIELIDATHNLGMCALV TRPC_HALVO : ARFVg.eDLPALVEAARDRGFQPLV TRPC_METTH : TGVFp..DLESGIETCRELSMEPLV Цветом выделены «биологически правильные» колонки – 22 шт. (88% от общего числа) Этот же участок выравнивания полученного с помощью программы ClustalW 140 TRPC_ACIAD TRPC_PHYPR TRPC_HALVO TRPC_METTH * 160 * 180 * : DPYNVVEARALQADCILLIVACLSDQQLEEMSKTAFEYDLDVLVEVHDEQELERALKLSEQCLLG : 190 : DVYQLLEARAYGADCVLLIVTLLSKEQLIELIDATHNLGMCALVEVNSVQELDIALAAKAR-LIG : 170 : NEAQLDVVQSD--LVLLIARFVG--EDLPALVEAARDRGFQPLVEVHTREELTAALAAGAD-IVG : 179 : DEYKIYQARASGASSVLLITGVF--PDLEAGIQKCRELSMEPLVECHTSLDIFRALEAGAE-IIG : 177 1 y 6 ara a 6L6i L LVEvh e6 AL a a 66G Участок выделен голубым цветом. Оранжевым цветом выделены аминокислоты, которыми данное выравнивание отличается от такового из BaliBase. Число совпавших колонок в выравниваниях составляет 18 (68% от общего числа «биологически правильных» колонок) Наблюдения Рассмотрим внимательно участки выравнивания последовательности TRPC_HALVO в двух выравниваниях, отличающиеся друг от друга. ARFVG--E (ClastalW) _ARFVg.e (BaliBase) Можно заметить, что в выравнивании программой ClustalW появился лишний геп (отмечен красным), из-за которого весь этот участок неправильный. Если его удалить, получим совпадающие выравнивания: ARFVG-E ARFVg.e Рассмотрим теперь в выравниваниях последовательности TRPC_METTH: TGVF--PDLEAGIQKCRELSMEPLV (ClastalW) TGVFp..DLESGIETCRELSMEPLV (BaliBase) Как видно они отличаются!! (обратите внимание на выделенное оранжевым). С помощью программы needle я построил выравнивание последовательности TRPC_METTH из банка данных SwissProt, на которую ссылается BaliBase в своем выравнивании, с последовательностью в самом выравнивании BaliBase… Результат оказался неожиданным: Identity 67%, Similarity 87.7% Из этого можно сделать вывод, что последовательность TRPC_METTH в банке Balibase и SwissProt не одна и та же…