Сравнение аминокислотных последовательностей белков и
реклама

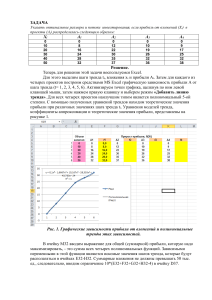
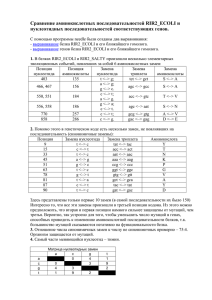
Сравнение аминокислотных последовательностей белков и нуклеотидных последовательностей соответствующих генов. Автор: Тихонов М.В. 201гр. Факультет биоинженерии и биоинформатики. МГУ им. Ломоносова. Результаты: 1) Создание выборки. Была получена выборка белков из банков SwissProt и UniProt. Затем по этим данным найдены соответствующие записи из банка EMBL (Coding Sequences). Полученные результаты приведены в таблице. 1 2 3 4 5 6 7 ID белка HMP_ECOLI Q3YZ03_SHISO HMP_SALCH HMP_ERWCT HMP_VIBCH HMP_DEIRA HMP_BACSH AC белка P24232 Q3YZ03 Q57LF5 Q6D245 Q9KMY3 Q9RYR5 P49852 BAA16460 AAZ89259 AAX66457 CAG76149 AAF96096 AAF12394 CAA05584 2) Наблюдение элементарных эволюционных событий в ближайших гомологах. В связи с отсутствием гомологов в диапазоне 97%, был выбран гомолог с идентичностью 99% (другой близкий гомолог был только с идентичностью 93%). Создано попарное выравнивание гомологичных аминокислотных и нуклеотидных последоваьельностей. * * 9 * * 18 * * 27 * * 36 * * 45 * g1 : atgcttgacgctcaaaccatcgctacagtaaaagccaccatccctttact : g2 : atgcttgacgctcaaaccatcgctacagtaaaagccaccatccctttact : 50 50 * 54 * * 63 * * 72 * * 81 * * 90 * * 99 g1 : ggtggaaacggggccaaagttaaccgcccatttctacgaccgtatgttta : g2 : ggtggaaacggggccaaagttaaccgcccatttctacgaccgtatgttta : 100 100 * *108 * *117 * *126 * *135 * *144 * * g1 : ctcataacccagaactcaaagaaatttttaacatgagtaaccagcgtaat : g2 : ctcataacccagaactcaaagaaatttttaacatgagtaaccagcgtaat : 150 150 153 * *162 * *171 * *180 * *189 * *198 g1 : ggcgatcaacgtgaagccctgtttaacgctattgccgcctacgccagtaa : g2 : ggcgatcaacgtgaagccctgtttaacgctattgccgcctacgccagtaa : 200 200 * *207 * *216 * *225 * *234 * *243 * *2 g1 : tattgaaaacctgcctgcgctgctgccagcggtagaaaaaatcgcgcaga : g2 : tattgaaaacctgcctgcgctgctgccagcggtagaaaaaatcgcgcaga : 250 250 52 * *261 * *270 * *279 * *288 * *297 * g1 : agcacaccagcttccagatcaaaccggaacagtacaacatcgtcggtgaa : g2 : agcacaccagcttccagatcaaaccggaacagtacaacatcgtcggtgaa : 300 300 *306 * *315 * *324 * *333 * *342 * *35 g1 : cacctgttggcaacgctggacgaaatgttcagcccggggcaggaagtgct : g2 : cacctgttggcaacgctggacgaaatgttcagcccggggcaggaagtgct : 350 350 1 * *360 * *369 * *378 * *387 * *396 * g1 : ggacgcgtggggtaaagcctatggtgtactggctaatgtatttatcaatc : 400 g2 : ggacgcgtggggtaaagcctatggtgtactggctaatgtatttatcaatc : 400 *405 * *414 * *423 * *432 * *441 * *450 g1 : gcgaggcggaaatctataacgaaaacgccagcaaagccggtggttgggaa : g2 : gcgaggcggaaatctataacgaaaacgccagcaaagccggtggttgggaa : 450 450 * *459 * *468 * *477 * *486 * *495 * g1 : ggtactcgcgatttccgcattgtggctaaaacaccgcgcagcgcgcttat : g2 : ggtactcgcgatttccgcattgtggctaaaacaccgcgcagcgcgcttat : 500 500 *504 * *513 * *522 * *531 * *540 * *549 g1 : caccagcttcgaactggagccggtcgacggtggcgcagtggcagaatacc : g2 : caccagcttcgaactggagccggtcgacggtggcgcagtggcagaatacc : 550 550 * *558 * *567 * *576 * *585 * *594 * * g1 : gtccggggcaatatctcggcgtctggctgaagccggaaggtttcccacat : g2 : gtccggggcaatatctcggcgtctggctgaagccggaaggtttcccgcat : 600 600 603 * *612 * *621 * *630 * *639 * *648 g1 : caggaaattcgtcagtactctttgactcgcaaaccggatggcaaaggcta : g2 : caggaaattcgtcagtactctttgactcgcaaaccggatggcaaaggcta : 650 650 * *657 * *666 * *675 * *684 * *693 * *7 g1 : tcgtattgcggtgaaacgcgaagagggtgggcaggtatccaactggttgc : g2 : tcgtattgcggtgaaacgcgaagagggtgggcaggtatccaactggttgc : 700 700 02 * *711 * *720 * *729 * *738 * *747 * g1 : acaatcacgccaatgttggcgatgtcgtgaaactggtcgctccggcaggt : g2 : acaatcacgccaatgttggcgatgtcgtgaaactggtcgctccggcaggt : 750 750 *756 * *765 * *774 * *783 * *792 * *80 g1 : gatttctttatggctgtcgcagatgacacaccagtgacgttaatctctgc : g2 : gatttctttatggctgtcgcagatgacacaccagtgacgttaatctctgc : 800 800 1 * *810 * *819 * *828 * *837 * *846 * g1 : cggtgttggtcaaacgccaatgctggcaatgctcgacacgctggcaaaag : g2 : cggtgttggtcaaacgccaatgctggcaatgctcgacacgctggcaaaag : 850 850 *855 * *864 * *873 * *882 * *891 * *900 g1 : caggccacacagcacaagtgaactggttccatgcggcagaaaatggcgat : g2 : caggccacacagcacaagtgaactggttccatgcggcagaaaatggcgat : 900 900 * *909 * *918 * *927 * *936 * *945 * g1 : gttcacgcctttgccgatgaagttaaggaactggggcagtcactgccgcg : g2 : gttcacgcctttgccgatgaagttaaggaactggggcagtcactgccgcg : 950 950 *954 * *963 * *972 * *981 * *990 * *999 g1 : ctttaccgcgcacacctggtatcgtcagccgagcgaagccgatcgcgcta : 1000 g2 : ctttaccgcgcacacctggcatcgtcagccgagcgaagccgatcgcgcta : 1000 * 1008 * 1017 * 1026 * 1035 * 1044 * 1 g1 : aaggtcagtttgatagcgaaggtctgatggatttgagcaaactggaaggt : 1050 g2 : aaggtcagtttgatagcgaaggtctgatggatttgagcaaactggaaggt : 1050 053 * 1062 * 1071 * 1080 * 1089 * 1098 g1 : gcgttcagcgatccgacaatgcagttctatctctgcggcccggttggctt : 1100 g2 : gcgttcagcgatccgacaatgcagttctatctatgcggcccggttggctt : 1100 * 1107 * 1116 * 1125 * 1134 * 1143 * 11 g1 : catgcagtttaccgcgaaacagttagtggatctgggcgtgaagcaggaaa : 1150 g2 : catgcagtttaccgcgaaacagttagtggatctgggcgtgaagcaggaaa : 1150 52 * 1161 * 1170 * 1179 * 1188 * g1 : acattcattacgaatgctttggcccgcataaggtgctgtaa : 1191 g2 : acattcattacgaatgctttggcccgcataaggtgctgtaa : 1191 ___________________ * 10 * 20 * 30 * 40 * 50 p1 : MLDAQTIATVKATIPLLVETGPKLTAHFYDRMFTHNPELKEIFNMSNQRN : p2 : MLDAQTIATVKATIPLLVETGPKLTAHFYDRMFTHNPELKEIFNMSNQRN : 50 50 * 60 * 70 * 80 * 90 * 100 p1 : GDQREALFNAIAAYASNIENLPALLPAVEKIAQKHTSFQIKPEQYNIVGE : 100 p2 : GDQREALFNAIAAYASNIENLPALLPAVEKIAQKHTSFQIKPEQYNIVGE : 100 * 110 * 120 * 130 * 140 * 150 p1 : HLLATLDEMFSPGQEVLDAWGKAYGVLANVFINREAEIYNENASKAGGWE : 150 p2 : HLLATLDEMFSPGQEVLDAWGKAYGVLANVFINREAEIYNENASKAGGWE : 150 * 160 * 170 * 180 * 190 * 200 p1 : GTRDFRIVAKTPRSALITSFELEPVDGGAVAEYRPGQYLGVWLKPEGFPH : 200 p2 : GTRDFRIVAKTPRSALITSFELEPVDGGAVAEYRPGQYLGVWLKPEGFPH : 200 * 210 * 220 * 230 * 240 * 250 p1 : QEIRQYSLTRKPDGKGYRIAVKREEGGQVSNWLHNHANVGDVVKLVAPAG : 250 p2 : QEIRQYSLTRKPDGKGYRIAVKREEGGQVSNWLHNHANVGDVVKLVAPAG : 250 * 260 * 270 * 280 * 290 * 300 p1 : DFFMAVADDTPVTLISAGVGQTPMLAMLDTLAKAGHTAQVNWFHAAENGD : 300 p2 : DFFMAVADDTPVTLISAGVGQTPMLAMLDTLAKAGHTAQVNWFHAAENGD : 300 * 310 * 320 * 330 * 340 * 350 p1 : VHAFADEVKELGQSLPRFTAHTWYRQPSEADRAKGQFDSEGLMDLSKLEG : 350 p2 : VHAFADEVKELGQSLPRFTAHTWHRQPSEADRAKGQFDSEGLMDLSKLEG : 350 * 360 * 370 * 380 * 390 * p1 : AFSDPTMQFYLCGPVGFMQFTAKQLVDLGVKQENIHYECFGPHKVL : 396 p2 : AFSDPTMQFYLCGPVGFMQFTAKQLVDLGVKQENIHYECFGPHKVL : 396 В аминокислотной последовательности найдена только одна замена по 324 позиции. Это соответствует замене по 970 позиции в нуклеотидной последовательности. Получается, что произошло одно из следующих событий событие: В исходном кодоне (неизвестно в каком именно TAT, CAT или может даже в другом) произошла мутация по первому из триплета нуклеотиду, что привело к изменению в аминокислотной последовательности. Кроме приведенной выше замены в ДНК есть еще две по 597 и 1083 позициям. Обе они произошли по третьей позиции кодона. В мутации по 597 позиции любая замена по третьей позиции будет синонимичной, так как любой кодон вида ССХ кодирует пролин. CCA ↕ CCG Р ↕ Р Во случае замены по 1083 позиции ситуация аналогична; любой триплет вида CTX кодирует лейцин. CTC ↕ CTA L ↕ L В данном случае соотношение синонимичных и несинонимичных замен нуклеотидов равно 2:1. Но это частный случай, и его нельзя считать общим для всех замен. Даже в таком случае видна основная закономерность стабилизирующего отбора – количество синонимичных (значит молчащих мутаций) больше, чем несинонимичных. Можно предположить, что если бы отличия в генах были бы более значительны, то отношение бы увеличилось в пользу синонимичных замен. Это легко объяснить тем, что белоки, как молекулы, выполняющая определенную функцию, имеет определенное строение, проверенное эволюцией и оптимальное для существования организма. Любая, не молчащая, мутация приводит к изменению структуры, и практически всегда нарушает функцию. Мутации же происходят и каждый раз они приводят к изменению или не изменения в белковой молекуле. Если мутация проявляется, как неблагоприятная то организм погибает, не оставив потомства. Если мутация не проявляется, то она может сохранится, что чаще всего и происходит. В другом случае, когда действует отбор на появление новых форм, отношение изменяется в противоположную сторону (соотношение синонимичных и несинонимичных замен нуклеотидов меньше 1). В данном типе отбора наоборот приветствуются несинонимичных мутации. Например, в вирусном антигене. Если антиген не накопит определенное количество несинонимичных мутаций, то его узнает иммунная система и уничтожит, в отличии от вирусов с измененным белком. Матрица замен нуклеотидов в таком виде практически не о чем не говорит – слишком мало данных. Можно предположить, что из-за равновероятности мутаций по каждому основанию, количество в каждой клетке должно быть приблизительно одинаковым. С другой стороны замена пурина на пурин или пиримидина на пиримидин (такие замены называются транзиции) более вероятна, чем замена пурин ↔ пиримидин (трансверсии). Даже в этой таблице с небольшим количеством данных видно, что более вероятны транзиции. Это можно объяснить объемным эффектом – пары, которые не только не комплиментарны, но и не подходят по размеру легко находятся репаративными ферментами. A A C G T C G 1 T 1 0 0 1 0 Какие данные можно получить из построенных графиков? (Графики находятся в файле excel graf.xls) Центральная линия тренда, соединяющая точки (100,100) и (5, 25) показывает идеализированную линейную модель замен, такую, где 100%-ое совпадение белков соответствует 100%-му соответствию генов, а если в белки идентичны на 5%, то ген – на 25% (это легко получить из вероятностных соображений). На самом деле такой расклад встречается довольно редко. Рассматривая графики зависимости процента идентичности гена от процента идентичности белка нужно, в первую очередь, обратить внимание на отклонение от линии тренда. Если график лежит значительно ниже линии тренда, что означает преобладание различий в гене от теоретического, то, скорей всего, в гене остаются молчащие мутации, не влияющие на структуру белка (например по 3-ей позиции). Это означает, что большинство проявляющихся мутаций негативно сказываются на белке и приводит к гибели. Значит, чем сильнее отклонился график вниз от линии тренда, тем сильнее давление отбора. Рассмотрим другой случай, когда график лежит выше линии тренда. Это означает, что белок поддается большему изменению, чем теоретический при одинаковом изменении ДНК. (По-моему, вполне вероятно, что сложится такая ситуация, когда любая замена нуклеотида приведет к замене в белке. В этом случае график будет лежать выше линии тренда.). Тогда график может лежать выше линии тренда.) Такое возможно, например, при дивергентной эволюции, смене функции, или преобладанием в белке аминокислот, для которых немного кодонов. Рассмотрим теперь конкретные графики. Видно, что оба графика, на большом протяжении лежат ниже, чем линия тренда. Это говорит о том, что белки консервативны, и замена аминокислоты ни к чему хорошему не приводит. В генах же мутации сохраняются, но опять же они – молчащие. В примере с вирусными белками материал богаче, поэтому график более вариабелен. Видно, в небольшой окрестности 90% есть локальный максимум. По всей вероятности, в этом диапазоне изменение белка не особенно влияет на его функции (например, меняется периферия белковой структуры). Что касается диапазона 82-98%, наблюдается локальный минимум. В этой позиции много молчащих мутаций гена. Далее при убывании id белка идет стабильное отклонение от линии тренда. Что касается графика гомологов hmp_ecoli, видно стабильное отклонение от линии тренда. Это говорит о стабильном давлении отбора. Сопроводительные материалы: файл graf.xls содержит графики. Файлы script_gen и script_pr содержат скрипт для попарного выравнивания соответственно белков и их генов.