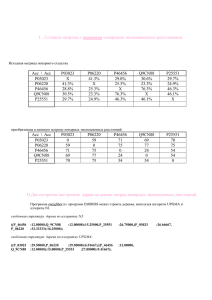
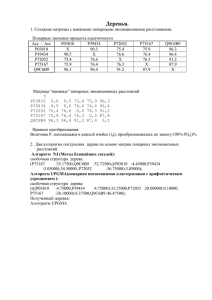
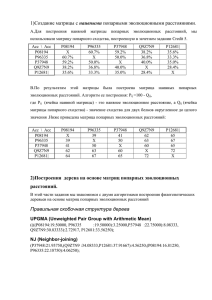
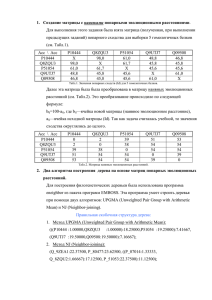
Отчет о проделанной работе по 27 занятию
реклама

Построение филогенетических деревьев 1. Создание матрицы с наивными попарными эволюционными расстояниями. При выполнении зачетного задания №5 мы строили матрицу попарного сходства для 5 белков: Acc \ Acc UBIC_ECOLI XYND_RUMFL TRYD_VIBCH ASNS_TRIVS ASNS_BRAOL UBIC_ECOLI X 4.3% 5.1% 5.8% 5.0% XYND_RUMFL 4.3% X 0.7% 4.3% 0.1% TRYD_VIBCH 5.1% 0.7% X 1.2% 0.1% ASNS_TRIVS 5.8% 4.3% 1.2% X 81.2% ASNS_BRAOL 5.0% 0.1% 0.1% 81.2% X По результатам этой матрицы была построена матрица наивных попарных эволюционных расстояний. Алгоритм ее построения можно записать так: Aij =100 - nij, где Aij – это наивное эволюционное расстояние, nij – значение сходства для двух белков (ID), округленное до целого значения (т.к. это учебная задача). Ниже приведена матрица с полученными результатами: Acc \ Acc UBIC_ECOLI XYND_RUMFL TRYD_VIBCH ASNS_TRIVS ASNS_BRAOL UBIC_ECOLI X 96 95 94 95 XYND_RUMFL 96 X 99 96 100 TRYD_VIBCH 95 99 X 99 100 ASNS_TRIVS 94 96 99 X 19 ASNS_BRAOL 95 100 100 19 X 2. Два алгоритма построения дерева на основе матриц попарных эволюционных расстояний. Для построения деревьев мы использовали программу eneighbor из пакета программ EBOSSUM. В этой части задания мы знакомимся с двумя алгоритмами построения филогенетических деревьев на основе матриц попарных эволюционных расстояний. Первый алгоритм – это Neighbor-joining, NJ (или алгоритм ближайших соседей). Второй - UPGMA (Unweighted Pair Group with Arithmetic Mean) или нечто вроде «попарной не взвешенной кластеризации с арифметическим усреднением». 3. Визуализация деревьев и сравнение их топологии. Филогенетическое дерево построенное по алгоритму NJ Филогенетическое дерево построенное по алгоритму UGMA Скобочные структуры: ((XYND_RUMFL:49.25000,(ASNS_TRIVS:8.50000,ASNS_BR AOL:10.50000):39.25000,(UBIC_ECOLI:45.50000, TRYD_VIBCH:49.50000):0.75000); (((UBIC_ECOLI:47.25000,(ASNS_TRIVS:9.50000 ,ASNS_BRAOL:9.50000):37.75000):1.41667, XYND_RUMFL:48.66667):0.45833,TRYD_VIBC H:49.12500); При построении филогенетических деревьев по различным алгоритмам получились различные результаты. Эволюция согласно дереву NJ: От какого-то гипотетического предка произошли три белка. Один из них – XYND_RUMFL, от второго произошли белки ASNS_TRIVS и ASNS_BRAOL, от третьего произошли: UBIC_ECOLI и TRYD_VIBCH Эволюция согласно дереву UPGMA: От гипотетического предка произошли два белка. Один из них –TRYD_VIBC. От другого произошли еще два белка: XYND_RUMFL и предок белков UBIC_ECOLI, ASNS_TRIVS и ASNS_BRAOL Отличие: Алгоритм NJ предполагает образование сразу трех «промежуточных» белков от древнего гипотетического предка, тогда как UPGMA рассматривает последовательное образование новых белков от предков в каждом поколении парами. Неукорененная кладограмма, на которой красным отмечен предполагаемый корень.