B L A
реклама

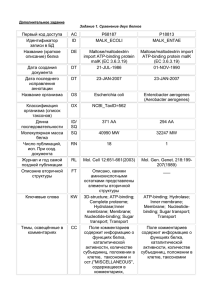
BLAST Задание №1. Создание индексных файлов для программ пакета BLAST Цель: создать индексные файлы пакета BLAST для поиска по геномам различныз бактерий Краткое описание работы: при выполнении задания были задействованы файлы pm_genome.fasta - полный геном бактерии Pasteurella multocida, pm_genome.fasta - полный геном бактерии Salmonella typhimurium, pm_genome.fasta - полный геном бактерии Xanthomonas campestris. Индексные файлы, требуемые в задании, были созданы с помощью команды formatdb. Используемые параметры: -i - файл, подаваемый на вход -p тип последовательности, записанной в файле (T - аминокислотная, F - нуклеотидная) -n название файлы (без расширения) Строка запроса в Unix: formatdb -i pm_genome.fasta -p F -n pm полученный файл лежит здесь. formatdb -i st_genome.fasta -p F -n st полученный файл лежит здесь. formatdb -i xc_genome.fasta -p F -n st полученный файл лежит здесь. Для поиска по трем геномам сразу была создана новая переменная: g_s="$genpath/st_genome.fasta $genpath/xc_genome.fasta $genpath/pm_genome.fasta". Строка запроса в Unix: formatdb -i $g_s.fasta -p F -n 3g полученный файл лежит здесь. Результаты: получены требуемые индексные файлы. Задание №2. Поиск в геноме участков, кодирующих белки, похожие на заданный Цель: выяснить, не закодированы ли белки, похожие на белок Malk_ecoli из организма Escherichia coli K-12 в неаннотированном геноме бактерий Pasteurella multocida, Salmonella typhimurium и Xanthomonas campestris. Краткое описание работы: для решения поставленной задачи была выбрана программа TBLASTN. Строка запроса в Unix: для Pasteurella multocida: blastall -p tblastn -d pm -i malk_ecoli.fasta -o pmres.txt для Salmonella typhimurium: blastall -p tblastn -d st -i malk_ecoli.fasta -o stres.txt для Xanthomonas campestris: blastall -p tblastn -d xc -i malk_ecoli.fasta -o xcres.txt Результаты: результаты сведены в Таблицу 1 Поиск гомологов Malk_ecoli Pasteurella multocida Salmonella typhimurium Xanthomonas campestris Число находок с 44 Е-value<0,001 55 32 AE006213 Pasteurella multocida subsp. multocida str. m70 section 180 of 204 of the complete genome. Характеристика Length = 10860 Score = лучшей находки: 290 bits (743) Expect = 7e-80 Identities = 164/354 (46%), Positives = 220/354 (62%), Gaps = 7/354 (1%) Frame = -1 AE008897| Salmonella typhimurium LT2, section 201 of 20 of the complete genome. Length = 20409 Score = 693 bits (1789), Expect = 0.0 Identities = 353/371 (95%), Positives = 364/371 (98%) Frame = +3 AE012320| Xanthomonas campestris pv. campestris str. ATCC 33913, section 228 of 460 of the complete genome. Length = 13110 Score = 320 bits (821), Expect = 1e-88 Identities = 182/363 (50%), Positives = 236/363 (65%), Gaps = 8/363 (2%) Frame = -1 E-value находки 7e-80 0.0 1e-88 AC соответствующей записи EMBL AE006213 AE008897 AE012320 Координаты выравнивания в записи EMBL 6846-5794 17748- 18854 6081- 5029 Координаты CDS complement(5740..6846) 17748..18857 complement(4987..6081) в записи EMBL AC UniProt в записи EMBL P19566 Q9CK71 Q8P8U9 Выводы: исходя из полученных результатов, можно сделать вывод, что во всех трех геномах есть белки, гомологичные белку Malk_ecoli из организма Escherichia coli K-12. В Таблице1 проанализирована первые из списков находок. Но всё же самым близким гомологом является - гомолог Malk_ecoli из организма Salmonella typhimurium выравнивание по длине равно 1106 остатков при e-value = 0.0 - довольно неплохой результат, если учесть, что мы искали в полном геноме. Задание №3. Аналогичный поиск сразу в нескольких геномах Цель: выяснить, не закодированы ли белки, похожие на белок Malk_ecoli из организма Escherichia coli K-12 в неаннотированных геномах бактерий Pasteurella multocida, Salmonella typhimurium или Xanthomonas campestris Краткое описание работы: индексные файлы для поиска по трем геномам сразу были созданы ранее (см. первое задание) С помощью программы TBLASTN был проведен поиск сразу по трем геномам бактерий Pasteurella multocida, Salmonella typhimurium и Xanthomonas campestris. Строка запроса в Unix: blastall -p tblastn -d 3g -i malk_ecoli.fasta -o 3gres.txt Результаты: результаты сведены в Таблице 2 . Количество находок с E-value<0.001 131 Характеристика лучшей находки AE008897| Salmonella typhimurium LT2, section 201 of 220 of the complete genome. Length = 20409 Score = 693 bits (1789), Expect = 0.0 Identities = 353/371 (95%), Positives = 364/371 (98%) Frame = +3 E-value лучшей находки 0.0 AC соответствующей записи EMBL AE008897 Координаты выравнивания в записи EMBL 17748- 18854 Координаты CDS в записи EMBL 17748..18857 AC UniProt в записи EMBL Q9CK71 Выводы: прежде всего стоит заметить, что во всех трех организмах достаточно много гомологов белка Malk_ecoli. Неудивительно, что суммарное количество находок с заданным e-value при поиске сначала по одному, потом по второму и третьему организму равно количеству находок при поиске в трех геномах сразу. Лучшей находкой как был, так и остался белок с АС=AE008897 из организма Salmonella typhimurium. В принципе это неудивительно, потому что значение e-value у этого белка минимальное - 0.0. Таким образом, эта находка по определению лучшая, поэтому она и осталась первой при поиске по трем геномам сразу. Задание №4. Поиск гомологов с помощью программы BLASTN Цель: поиск гомологов определенного гена белка Malk_ecoli из организма Escherichia coli с помощью программы BLASTN. Краткое описание работы: использовался файл gen_malk.fasta с геном белка Malk_ecoli. Поиск гомологов во всех трех геномах (pm_genome.fasta, st_genome.fasta и xc_genome.fasta) был произведен следующим образом: blastall -p blastn -d 3g -i gen_malk.fasta -o MALK_ECOLI.txt Результаты: Описание лучшей находки. Выводы: Наверное, главным наблюдением можно считать, что разброс E-value и Score меньше при поиске с помощью программы TBLASTN, нежели с помощью программы BLASTN. Это свидетельствует о том, что BLASTN хорошо подходит для поиска самых близких гомологов, ас помощью TBLASTN можно искать и не самые близкие гомологи. Это связано со многими параметрами, которые лежат в основе работы обеих программ. В принципе работы TBLASTN лежит поиск по аминокислотной последовательности с учетом нескольких соответствующих нуклеотидных последовательностей (вырожденность генетического кода, чаще всего в 3 позиции). А в BLASTN такого нет. Поэтому получили больше гомологов с несильно различающимся и достаточно маленьким значением e-value. Все эти факты на практике подтвердили то, что мы уже знаем в теории - TBLASTN подходит для поиска всех гомологов (с разной степенью гомологии, не обязательно очень близкие). А BLASTN подходит для поиска очень близких гомологов.