Элементарная эволюционная модель
реклама

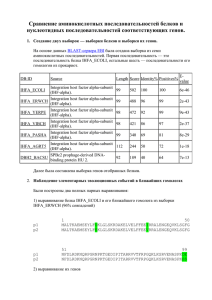
Cравнение аминокислотных последовательностей белков и нуклеотидных последовательностей соответствующих генов Создание двух выборок — выборки белков и выборки их генов с помощью BLAST-сервера EBI. Белок (AC) p1 (P09373_ECOLI) p2 (Q83LP6_SHIFL) p3 (Q57R28_SALCH) p4 (Q7N6E2_PHOLL) p5 (Q9S4V8_AERHY) p6 (Q89YJ3_BACTN) p7 (Q92A91_LISIN) Ген (AC) g1 (AAC73989) g2 (AAN42528) g3 (AAX64833) g4 (CAE13906) g5 (AAD52103) g6 (AAO79843) g7 (CAC97261) Наблюдение элементарных эволюционных событий в ближайших гомологах 1. При выравнивании белка PFLB_ECOLI и его ближайшего гомолога Q83LP6_SHIFL (~97% совпадений) программой needle обнаружено пять аминокислотных замен: в 93-ой позиции – Q/A, в 97-ой позиции – I/V, в 198-ой позиции – L/Y, в 470-ой позиции – V/L, в 472-ой позиции – N/S. Так выглядит содержащая замены: часть выравнивания белковых последовательностей, * 100 * 120 * 140 * 160 * 1 p1 : INKQLEKIVGLQTEAPLKRALIPFGGIKMIEGSCKAYNRELDPMIKKIFTEYRKTHNQGVFDVYTPDILRCRKSGVLTGLPDAYGRGRI : 177 p2 : INKALEKVVGLQTEAPLKRALIPFGGIKMIEGSCKAYNRELDPMIKKIFTEYRKTHNQGVFDVYTPDILRCRKSGVLTGLPDAYGRGRI : 178 80 * 200 * 220 * 240 * 260 p1 : IGDYRRVALYGIDYLMKDKLAQFTSLQADLENGVNLEQTIRLREEIAEQHRALGQMKEMAAKYGYDISGPATNAQEAIQWTYFGYLAAV : 266 p2 : IGDYRRVALYGIDYLMKDKYAQFTSLQADLENGVNLEQTIRLREEIAEQHRALGQMKEMAAKYGYDISGPATNAQEAIQWTYFGYLAAV : 267 * 280 * 300 * 320 * 340 * p1 : KSQNGAAMSFGRTSTFLDVYIERDLKAGKITEQEAQEMVDHLVMKLRMVRFLRTPEYDELFSGDPIWATESIGGMGLDGRTLVTKNSFR : 355 p2 : KSQNGAAMSFGRTSTFLDVYIERDLKAGKITEQEAQEMVDHLVMKLRMVRFLRTPEYDELFSGDPIWATESIGGMGLDGRTLVTKNSFR : 356 360 * 380 * 400 * 420 * 440 p1 : FLNTLYTMGPSPEPNMTILWSEKLPLNFKKFAAKVSIDTSSLQYENDDLMRPDFNNDDYAIACCVSPMIVGKQMQFFGARANLAKTMLY : 444 p2 : FLNTLYTMGPSPEPNMTILWSEKLPLNFKKFAAKVSIDTSSLQYENDDLMRPDFNNDDYAIACCVSPMIVGKQMQFFGARANLAKTMLY : 445 * 460 * 480 * 500 * 520 * p1 : AINGGVDEKLKMQVGPKSEPIKGDVLNYDEVMERMDHFMDWLAKQYITALNIIHYMHDKYSYEASLMALHDRDVIRTMACGIAGLSVAA : 533 p2 : AINGGVDEKLKMQVGPKSEPIKGDLLSYDEVMERMDHFMDWLAKQYITALNIIHYMHDKYSYEASLMALHDRDVIRTMACGIAGLSVAA : 534 Лиловым выделены замены. 2. Какие элементарные эволюционные события повлекли за собой аминокислотные замены? Так выглядит часть выравнивания нуклеотидных последовательностей, содержащая нуклеотидные замены: * 160 * 180 * 200 * g1 : ctgaagcgaccaccaccctgtgggacaaagtaatggaaggcgttaaactggaaaaccgcactcacgcgcca : g2 : ctgaagcgaccaccaccctgtgggacaaagtaatggaaggtgttaaactggaaaaccgcactcacgcgcca : 213 213 220 * 240 * 260 * 280 g1 : gttgactttgacaccgctgttgcttccaccatcacctctcacgacgctggctacatcaacaagcagc-ttg : g2 : gttgattttgacaccgctgttgcttccaccatcacctctcacgacgctggctacatcaataa--agcgttg : 283 282 * 300 * 320 * 340 * g1 : agaaaa--tcgttggtctgcagactgaagctccgctgaaacgtgctcttatcccgttcggtggtatcaaaa : g2 : -gaaaaagttgttggtctgcagactgaagctccgctgaaacgtgctcttatcccgttcggtggtatcaaaa : 352 352 360 * 380 * 400 * 420 g1 : tgatcgaaggttcctgcaaagcgtacaaccgcgaactggatccgatgatcaaaaaaatcttcactgaatac : g2 : tgatcgaaggttcctgcaaagcgtacaaccgcgaactggacccgatgatcaaaaaaatcttcactgaatac : 423 423 * 440 * 460 * 480 * g1 : cgtaaaactcacaaccagggcgtgttcgacgtttacactccggacatcctgcgttgccgtaaatctggtgt : g2 : cgtaaaactcacaaccagggcgtgttcgacgtttacactccggacatcctgcgttgccgtaaatccggtgt : 494 494 500 * 520 * 540 * 560 g1 : tctgaccggtctgccagatgcatatggccgtggccgtatcatcggtgactaccgtcgcgttgcgctgtacg : g2 : tctgaccggtctgccagatgcttatggccgtggccgtatcatcggtgactaccgtcgcgttgcgctgtacg : 565 565 * 580 * 600 * 620 * 64 g1 : gtatcgactacctgatgaaagacaaactggcacagttcacttctctgcaggctgatctggaaaacggcgta : g2 : gtatcgactacctgatgaaagacaaatacgctcagttcacctctctacaggctgatctggaaaacggcgta : 636 636 0 * 660 * 680 * 700 * g1 : aacctggaacagactatccgtctgcgcgaagaaatcgctgaacagcaccgcgctctgggtcagatgaaaga : g2 : aacctggaacagactatccgtctgcgcgaagaaatcgctgaacagcaccgcgctctgggtcagatgaaaga : 707 707 720 * 740 * 760 * 780 g1 : aatggctgcgaaatacggctacgacatctctggtccggctaccaacgctcaggaagctatccagtggactt : g2 : aatggctgcgaaatacggctacgacatctctggtccggctaccaacgctcaggaagctatccagtggactt : 778 778 * 800 * 820 * 840 * g1 : acttcggctacctggctgctgttaagtctcagaacggtgctgcaatgtccttcggtcgtacctccaccttc : g2 : acttcggctacctggctgctgttaagtctcagaacggtgctgcaatgtccttcggtcgtacctccaccttc : 849 849 860 * 880 * 900 * 920 g1 : ctggatgtgtacatcgaacgtgacctgaaagctggcaagatcaccgaacaagaagcgcaggaaatggttga : g2 : ctggatgtgtacatcgaacgtgacctgaaagctggcaagatcaccgaacaagaagcgcaggaaatggttga : 920 920 * 940 * 960 * 980 * g1 : ccacctggtcatgaaactgcgtatggttcgcttcctgcgtactccggaatacgatgaactgttctctggcg : g2 : ccacctggtcatgaaactgcgtatggttcgcttcctgcgtactccggaatacgatgaactgttctctggcg : 991 991 1000 * 1020 * 1040 * 1060 g1 : acccgatctgggcaaccgaatctatcggtggtatgggcctcgacggtcgtaccctggttaccaaaaacagc : 1062 g2 : acccaatctgggcaaccgaatctatcggtggtatgggcctcgatggtcgtaccctggttaccaaaaacagc : 1062 * 1080 * 1100 * 1120 * g1 : ttccgtttcctgaacaccctgtacaccatgggtccgtctccggaaccgaacatgaccattctgtggtctga : 1133 g2 : ttccgtttcctgaacaccctgtacactatggggccgtctccggaaccgaacatgaccattctgtggtctga : 1133 1140 * 1160 * 1180 * 1200 g1 : aaaactgccgctgaacttcaagaaattcgccgctaaagtgtccatcgacacctcttctctgcagtatgaga : 1204 g2 : aaaactgccgctgaacttcaagaaattcgccgctaaagtgtccatcgacacctcttctctgcagtatgaga : 1204 * 1220 * 1240 * 1260 * 12 g1 : acgatgacctgatgcgtccggacttcaacaacgatgactacgctattgcttgctgcgtaagcccgatgatc : 1275 g2 : acgatgacctgatgcgtccggacttcaacaacgatgactacgctatcgcttgctgcgtaagcccgatgatc : 1275 80 * 1300 * 1320 * 1340 g1 : gttggtaaacaaatgcagttcttcggtgcgcgtgcaaacctggcgaaaaccatgctgtacgcaatcaacgg : 1346 g2 : gttggtaaacaaatgcagttcttcggtgcgcgtgcaaacctggcgaaaaccatgctgtacgcaatcaacgg : 1346 * 1360 * 1380 * 1400 * 1420 g1 : cggcgttgacgaaaaactgaaaatgcaggttggtccgaagtctgaaccgatcaaaggcgatgtcctgaact : 1417 g2 : cggcgttgacgaaaaactgaaaatgcaggttggtccgaagtctgaaccgatcaaaggcgatctcctgagct : 1417 * 1440 * 1460 * 1480 * g1 : atgatgaagtgatggagcgcatggatcacttcatggactggctggctaaacagtacatcactgcactgaac : 1488 g2 : acgatgaagtgatggagcgcatggatcacttcatggactggctggctaaacagtacatcactgcactgaac : 1488 1500 * 1520 * 1540 * 1560 g1 : atcatccactacatgcacgacaagtacagctacgaagcctctctgatggcgctgcacgaccgtgacgttat : 1559 g2 : atcatccactacatgcacgacaagtacagctacgaagcctctctgatggcgctgcacgaccgtgacgttat : 1559 * 1580 * 1600 * 1620 * g1 : ccgcaccatggcgtgtggtatcgctggtctgtccgttgctgctgactccctgtctgcaatcaaatatgcga : 1630 g2 : ccgcaccatggcgtgtggtatcgctggtctgtccgttgctgctgactccctgtctgcaatcaaatatgcga : 1630 1640 * 1660 * 1680 * 1700 g1 : aagttaaaccgattcgtgacgaagacggtctggctatcgacttcgaaatcgaaggcgaatacccgcagttt : 1701 g2 : aagttaaaccgattcgtgacgaagacggtctggctatcgacttcgaaatcgaaggcgaatacccgcagttt : 1701 * 1720 * 1740 * 1760 * g1 : ggtaacaatgatccgcgtgtagatgacctggctgttgacctggtagaacgtttcatgaagaaaattcagaa : 1772 g2 : ggtaacaatgatccgcgtgtagatgacctggctgttgacctggtagaacgtttcatgaagaaaattcagaa : 1772 1780 * 1800 * 1820 * 1840 g1 : actgcacacctaccgtgacgctatcccgactcagtctgttctgaccatcacttctaacgttgtgtatggta : 1843 g2 : actgcacacctaccgtgacgctatcccgactcagtctgttctgaccatcacttctaacgttgtgtatggta : 1843 * 1860 * 1880 * 1900 * 1 g1 : agaaaacgggtaacaccccagacggtcgtcgtgctggcgcgccgttcggaccgggtgctaacccgatgcac : 1914 g2 : agaaaactggtaacaccccagacggtcgtcgtgctggcgcgccgttcggaccgggtgctaacccgatgcac : 1914 920 * 1940 * 1960 * 1980 g1 : ggtcgtgaccagaaaggtgcagtagcctctctgacttccgttgctaaactgccgtttgcttacgctaaaga : 1985 g2 : ggtcgtgaccagaaaggtgctgtagcgtctctgacttccgttgctaaactgccgtttgcttacgctaaaga : 1985 Голубым выделены делеции. Лиловым выделены замены в третьей позиции. Серым выделены замены в первой и второй позициях. В рамочку обведены замены в нуклеотидной последовательности, повлекшие изменения в аминокислотной последовательности: 1) Q – A (276-281 позиции): две несинонимичные замены - по первой позиции С G, и по второй позиции А - С. GCAGC--AGCG 2) L – Y (595-597 позиции): две несинонимичные замены - по первой позиции С T, и по второй позиции Т - А. CTG TAC 3) N – S (1417-1419 позиции): одна несинонимичная замена по второй позиции А G. AAC AGC 4) I – V (289-291 позиции): одна несинонимичная замена по первой позиции А - G. A--TC AAGTT 5) V – L (1408-1410 позиции): одна несинонимичная замена по первой позиции G – C. GTC CTC Отметим следующую особенность выравнивания: нуклеотидная последовательность, соответствующая первой и второй аминокислотным заменам, была выровнена следующим образом: gcagc-ttgagaaaa--tc --agcgttg-gaaaaagtt Как видно, появляются делеции. Но возможен другой вариант выравнивания: gcagcttgagaaaatc agcgttggaaaaagtt Такое выравнивание полностью верно, но предполагается наличие большего числа замен в последовательностях. Заметим, что имеющаяся замена пятого нуклеотида данного выравнивания C/T, хотя и произошла по «первой позиции», является синонимичной. Случаев синонимичных замен по первоначальному выравниванию: 20. Из них в третьих позициях кодонов: 20. Случаев синонимичных замен по выравниванию без делеций: 24. Из них в третьих позициях кодонов: 23. Соотношение между синонимичными и несинонимичными заменами по выравниванию, не содержащему делеции: 24/7 ~ 3,4 Чем чаще происходит синонимичная замена по сравнению с несинонимичной, тем более консервативная последовательность. Довольно высокое значение отношения свидетельствует о консервативности. «Матрица замен» нуклеотидов: a c g t a 0 c g 0 3 t 4 3 4 1 2 2 3 3 1 2 2 Известна закономерность: транзиции более вероятны, чем трансверсии. То есть, вероятнее замена пуринового основания на пуриновое, чем на пиримидиновое. Из этого следует, что замены A/G и C/T встречаются чаще. По получившейся матрице замен эта закономерность хорошо выявляется для пары C/T, а неочевидность преобладания замены A/G можно списать на недостаточность данных. Исследование зависимости процента совпадений последовательностей белков от процента совпадений последовательностей их генов Haem agglutinin 100 Gene % identity 80 60 40 20 0 0 20 40 60 80 100 Protein % identity График 1. Пунктирная линия (ось С) показывает зависимость процента совпадений в белковых последовательностях от совпадений в нуклеотидной последовательности. Ось С получена следующим образом: прямой соединены точки, отвечающие за выравнивание, с одной стороны, последовательности некоторого белка с самим собой (100, 100), с другой стороны, последовательностей двух несвязанных белков (5, 25). Синяя ломаная – аналогичная зависимость для выборки гомологов белка гемагглютинина. Formate acetyltransferase 1 100% Gene % identity 80% 60% 40% 20% 0% 0% 20% 40% 60% 80% 100% Protein % identity График 2. Розовая линия (ось С) показывает зависимость процента совпадений в белковых последовательностях от совпадений в нуклеотидной последовательности. Ось С получена следующим образом: прямой соединены точки, отвечающие за выравнивание, с одной стороны, последовательности некоторого белка с самим собой (100, 100), с другой стороны, последовательностей двух несвязанных белков (5, 25). Синяя ломаная – аналогичная зависимость для выборки гомологов белка форматацетилтрансферазы 1. На первый взгляд графики похожи, но стоит отметить отклонение графика форматацетилтрансферазы 1 от оси С по сравнению с графиком гемагглютинина. Известно, что вирус гриппа распространяется среди людей, присоединяясь к человеческим клеткам с помощью белка гемагглютинина, находящегося на оболочке вируса. Поскольку конфигурация аминокислот, определяющих разновидность белка гемагглютинина, часто изменяется, ежегодно появляется новый тип вируса гриппа. Из этих данных можно сделать вывод, что первый график соответствует набору мало консервативных последовательностей. В то же время было получено, что гомологи белка форматацетилтрансферазы 1 являются довольно консервативными. Этот факт подтверждается соответствующим графиком. Графий, (по крайней мере, для двух ближайших гомологов, для которых вычислено отношение синонимичных и несинонимичных замен) форматацетилтрансферазы 1 сильнее отклоняется от оси С, в то время как для гемагглютинина этого не наблюдается.