13 gamma proteo bacteria
реклама
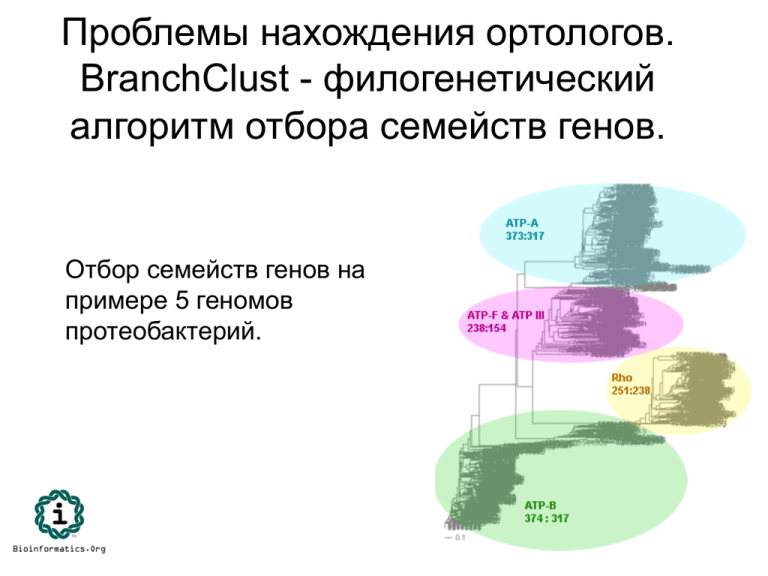
Проблемы нахождения ортологов.
BranchClust - филогенетический
алгоритм отбора семейств генов.
Отбор семейств генов на
примере 5 геномов
протеобактерий.
Бесплатный SCP-клиент
BranchClust - филогенетический алгоритм
отбора семейств генов
Семейство АТФ-синтаз
Случай 2-х бактерий и 2-х архей
ATP-A (catalytic subunit)
Escherichia coli
Escherichia coli
ATP-A
ATP-B
ATP-B (non-catalytic subunit)
ATP-A
ATP-B
Methanosarcina
mazei
Methanosarcina
mazei
ATP-F
ATP-A
ATP-A
ATP-A
ATP-A
ATP-B
ATP-B
ATP-B
ATP-B
Sulfolobus
solfataricus
Sulfolobus
solfataricus
ATP-A
ATP-B
Bacillus subtilis
ATP-A
ATP-B
ATP-F
Bacillus subtilis
Метод RBH не отбирает ни ATP-A, ни ATB-B
Families of ATP-synthases
Phylogenetic Tree
Family of ATP-A
Sulfolobus solfataricus
ATP-A
Methanosarcina
mazei
ATP-A
Bacillus subtilis
ATP-A
ATP-A
Escherichia coli
Bacillus subtilis ATP-F
ATP-B
Escherichia coli
Escherichia coli
ATP-F
ATP-B
ATP-B
Family of ATP-F
Sulfolobus
solfataricus
Bacillus subtilis
ATP-B
Methanosarcina
mazei
Family of ATP-B
BranchClust Algorithm
genome 1
genome 2
BLAST genome 3
hits
genome i
genome N
dataset of N genomes
www.bioinformatics.org/branchclust
superfamily
tree
BranchClust Algorithm
www.bioinformatics.org/branchclust
BranchClust Algorithm
Root positions
Superfamily of penicillin-binding protein
13 gamma proteo bacteria
www.bioinformatics.org/branchclust
Superfamily of DNA-binding protein
13 gamma proteo bacteria
Пример: 5 протеобактерий
1 Gamma-proteobacteria
Escherichia_coli_K_12_substr__MG1655_uid57779
2 Beta-proteobacteria
Bordetella_parapertussis_12822_uid57615
3 Alpha-proteobacteria
Rickettsia_prowazekii_Madrid_E_uid61565
4 Epsilon-proteobacteria
Helicobacter_pylori_26695_uid57787
5 Delta-proteobacteria
Desulfovibrio_vulgaris_DP4_uid58679
QuickTime™ and a
TIFF (Uncompressed) decompressor
are needed to see this picture.
Дерево
построено
конкатенацией
ортологов из 31
семейства из
191 видов
Ciccarelli et al. 2006, Science
Редактируем файл
.bash_profile
>cd
>vi .bash_profile
export PATH=/usr/local/biotools/bin:/usr/local/biotools/data/:$PATH
export BLASTMAT=/usr/local/biotools/data/
Устанавливаем BioPerl
Копируем пакет bioperl в домашнюю директорию
Создать один файл,
содержащий все 5 геномов
>perl create_one_faa.pl
#!usr/bin/perl -w
#create dir
if (!opendir(DIR,"")){
mkdir("fasta_all");
}else{
close(DIR);
}
system(" > fasta_all/allgenomes.faa");
while(defined($file=glob("fasta/*.faa")))
{
system("cat $file >> fasta_all/allgenomes.faa");
}
Форматировать файл для
использования blast
>perl format_faa.pl
#! /usr/bin/perl -w
system("formatdb -o T -p T -i
fasta_all/allgenomes.faa");
bio568b-2:test mariap$ ls fasta_all
allgenomes.faa
allgenomes.faa.pin
allgenomes.faa.pni
allgenomes.faa.psi
allgenomes.faa.phr
allgenomes.faa.pnd
allgenomes.faa.psd
allgenomes.faa.psq
Запускаем программу BLAST
Примерное время работы для нашего файла - 30 минутт
(когда запущен один вариант программы)
>perl do_blast.pl
#!/usr/bin/perl -w
#create 'blast' dir if it doesn't exist
if (!opendir(DIR,"blast")){
mkdir("blast");
}else{
close(DIR);
}
$blast_input="fasta_all/allgenomes.faa";
$blast_output="blast/all_vs_all.out";
#system("blastall -i $blast_input -d $blast_input -p blastp -o
$blast_output -I T -e 1E-4 -F F -W 2 -m 8");
system("blastall -i $blast_input -d $blast_input -p blastp -o
$blast_output -I T -e 1E-4 -m 8");
Отслеживаем свои процессы
>perl do_blast.pl
- убить ctrl-c
>perl do_blast.pl & (запуск на background)
Просмотр процессов
>ps
>ps aux
>top
Убить свои процессы
>kill -9 pid
где pid - номер процесса
Результаты blast
> more blast/all_vs_all.out | wc -l
221973
more blast/all_vs_all.out
Обрабатываем результаты blast
> more blast/all_vs_all.out | wc -l
221973
my $in = new Bio::SearchIO(-format => 'blasttable',
-file => "$infile");
> perl parse_blast.pl
# Because it is blast of a database of N genomes
against itself,
# first hit for each gene is the gene itself.
# That is why we assemble only hits.
#! /usr/bin/perl -w
use lib "/Users/mariap/bioperl-1.5-my";
use Bio::SearchIO;
#create 'parsed' if it doesn't exist
if(!opendir(DIR,"parsed")){
mkdir("parsed");
}else{
close(DIR);
}
$infile="blast/all_vs_all.out";
$outfile="parsed/all_vs_all.parsed";
open (OUT, ">$outfile") || die "Cannot open file
$outfile $!\n";
while(my $result = $in->next_result){
while($hit = $result->next_hit()){
# take only first hsp for every hit, it has the best
e-value
# exctract gene number
$hit->name()=~/\|(.+?)\|/s;
$gene=$1;
print OUT "$gene\t";
}
print OUT "\n";
}
close (OUT);
Обрабатываем результаты blast
>more parsed/all_vs_all.parsed
Идентификация видов по номерам
gi (gene identification)
>perl extract_gi_numbers.pl
>more gi_numbers.out
Bordetella parapertussis 12822 |
3359....
1616.....
Desulfovibrio vulgaris DP4 | 1206.....
162139...
Escherichia coli str. K-12 substr. MG1655 | 1612....
4917....
9011....
2218.....
2265.....
1456.....
9454....
3454.....
1577.....
3082.....
2885.....
1717.....
1613....
6700....
3334....
2960.....
162135...
Helicobacter pylori 26695 | 1564....
1613.....
4873....
Rickettsia prowazekii str. Madrid E | 1560....
1617.....
Отбираем суперсемейства,
содержащие, по крайней мере, 4 вида.
>perl parse_superfamilies_singlelink.pl 5 &
Результат:
> more parsed/all_vs_all.fam | wc -l
604
>perl simple_info.pl parsed/all_vs_all.fam
Результат: parsed/all_vs_all.fam.info
>perl sort_column.pl parsed/all_vs_all.fam.info
Результат: parsed/all_vs_all.fam.info.sorted
Отбираем последовательности
для суперсемейств
>perl prepare_fa.pl parsed/all_vs_all.fam
Результат:
fa/fam_XX.fa
Посмотреть содержимое:
>less fa/fam_7.fa
Проверить, что число найденных последовательностей совпадает с числом
генов в суперсемействе:
>more fa/fam_12.fa | grep '>' | wc -l
599
Выравнивание суперсемейств
~10 минут без больших суперсемейств
Уберем большие суперсемейства в другую директорию
>mkdir fa_big
>mv fa/fam_12.fa fa_big/
> mv fa/fam_52.fa fa_big/
>mv fa/fam_98.fa fa_big/
>mv fa/fam_57.fa fa_big/
>mv fa/fam_58.fa fa_big/
>mv fa/fam_60.fa fa_big/
Запустим выравнивание
>perl do_clustalw_aln.pl &
Результат:
dist/*.aln
#! /usr/bin/perl -w
#create 'dist' if it doesn't exist
if (!opendir(DIR,"dist")){
mkdir("dist");
}else{
close(DIR);
}
while(defined($filename=glob("fa/*.fa"))
)
{
print "$filename\n";
# clustalw each file
system("clustalw -infile=$filename align -type=protein");
}
system("mv fa/*.aln dist/$d");
Построение деревьев методом
расстояний с коррекцией Кимуры
>do_clustalw_dist_kimura.pl
Результат: dist/fam_##.ph
Подготовка деревьев для
BranchClust:
>perl prepare_trees.pl
Результат:
trees/fam_##.tre
#! /usr/bin/perl -w
# Tree reconstruction, using
distance method with kimura
correction
# trees will be generated in the
same directory 'dist' with
extension *.ph
while(defined($filename=glob("dist/
*.aln")))
{
# clustalw each file
system("clustalw -infile=$filename
-tree -OUTPUTTREE=dist -kimura");
}
Обработка деревьев
суперсемейств алгоритмом
BranchClust
> perl branchclust_all.pl 4 &
Результаты: clusters/clusters_##.out
clusters/family_##.
Дополнительно: clusters_##.log
>perl names_for_cluster_all.pl
>perl detailed_summary.pl
Результаты:
detailed_summary.out
families-names.list
detailed_summary.out
tRNA synthases
>more clusters/clusters_148.out.names
ATP-synthases