Зачетное задание по 1 блоку 2 семестра
реклама

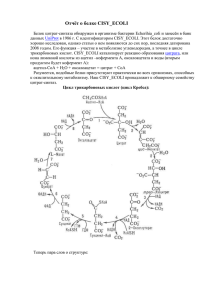
Зачетное задание по 1 блоку 2 семестра Нагаев Борис Коды моего белка: LPP_ECOLI P69776 1JCC Ссылки на предыдущие работы, посвященные белку: http://kodomo.cmm.msu.ru/~bnagaev/lpp_ecoli/ – все работы, связанные с белком http://kodomo.cmm.msu.ru/~bnagaev/lpp_ecoli/about_protein/ – общие сведения о белке http://kodomo.cmm.msu.ru/~bnagaev/lpp_ecoli/pdb/ – исследование структуры Название моего белка: главный липопротеин наружной мембраны. Функционирующий белок является тримером: он состоит из трёх одинаковых субъединиц, контактирующих своими гидрофобными частями. Каждая субъединица содержит альфаспираль длиной 47 аминокислотных остатков (с 25 до 71). Аминокислотные остатки с 1 до 20 являются сигнальной последовательностью. Сигнальные последовательности свойственны белкам, предназначенным ЭПР. Впоследствии такие белки секретируются внеклеточно или в периплазму. Обычно сигнальные последовательности удаляются у зрелых белков. Масса одной зрелой цепи составляет 8287 Да. Последовательность белка в fasta-формате: >sp|P69776|LPP_ECOLI Major outer membrane lipoprotein OS=Escherichia coli (strain K12) GN=lpp PE=1 SV=1 MKATKLVLGAVILGSTLLAGCSSNAKIDQLSSDVQTLNAKVDQLSNDVNAMRSDVQAAKD DAARANQRLDNMATKYRK Эта последовательность размещена на сервере UniProt по адресу: http://www.uniprot.org/uniprot/P69776.fasta Белок располагается на наружной мембране клеток кишечной палочки, где он играет роль липидного и пептидоглюканового якоря. Он взаимодействует с пептидоглюканами как ковалентно, так и нековалентно. Это взаимодействие способствует обеспечению структурной и функциональной целостности клеточной стенки. Организм: Escherichia coli (strain K12). Полная систематика: Bacteria › Proteobacteria › Gammaproteobacteria › Enterobacteriales › Enterobacteriaceae › Escherichia В пространстве белок складывается в такую структуру: Одна цепочка в ленточной модели выглядит следующим образом: Эти изображения показывают, что основная часть белка представлена альфа-спиралью. Ссылки на записи про белок: UniProt: http://www.uniprot.org/uniprot/P69776.txt – исходный текст записи UniProt http://www.uniprot.org/uniprot/P69776 – дружественный интерфейс UniProt PDB: http://www.rcsb.org/pdb/files/1jcc.pdb – исходный текст записи PDB http://www.rcsb.org/pdb/explore/explore.do?structureId=1JCC – дружественный интерфейс Статьи про белок: "DNA sequence of the gene for the outer membrane lipoprotein of E. coli: an extremely AT-rich promoter." Nakamura K., Inouye M. Cell 18:1109-1117(1979) [PubMed: 391404] [Abstract] Cited for: NUCLEOTIDE SEQUENCE [GENOMIC DNA]. Strain: B. "Messenger ribonucleic acid of the lipoprotein of the Escherichia coli outer membrane. II. The complete nucleotide sequence." Nakamura K., Pirtle R.M., Pirtle I.L., Takeishi K., Inouye M. J. Biol. Chem. 255:210-216(1980) [PubMed: 6765942] [Abstract] Cited for: NUCLEOTIDE SEQUENCE [GENOMIC DNA]. "A 570-kb DNA sequence of the Escherichia coli K-12 genome corresponding to the 28.040.1 min region on the linkage map." Aiba H., Baba T., Fujita K., Hayashi K., Inada T., Isono K., Itoh T., Kasai H., Kashimoto K., Kimura S., Kitakawa M., Kitagawa M., Makino K., Miki T., Mizobuchi K., Mori H., Mori T., Motomura K. Horiuchi T. DNA Res. 3:363-377(1996) [PubMed: 9097039] [Abstract] Cited for: NUCLEOTIDE SEQUENCE [LARGE SCALE GENOMIC DNA]. Strain: K12 / W3110 / ATCC 27325 / DSM 5911. "The complete genome sequence of Escherichia coli K-12." Blattner F.R., Plunkett G. III, Bloch C.A., Perna N.T., Burland V., Riley M., Collado-Vides J., Glasner J.D., Rode C.K., Mayhew G.F., Gregor J., Davis N.W., Kirkpatrick H.A., Goeden M.A., Rose D.J., Mau B., Shao Y. Science 277:1453-1474(1997) [PubMed: 9278503] [Abstract] Cited for: NUCLEOTIDE SEQUENCE [LARGE SCALE GENOMIC DNA]. Strain: K12 / MG1655 / ATCC 47076. "Highly accurate genome sequences of Escherichia coli K-12 strains MG1655 and W3110." Hayashi K., Morooka N., Yamamoto Y., Fujita K., Isono K., Choi S., Ohtsubo E., Baba T., Wanner B.L., Mori H., Horiuchi T. Mol. Syst. Biol. 2:E1-E5(2006) [PubMed: 16738553] [Abstract] Cited for: NUCLEOTIDE SEQUENCE [LARGE SCALE GENOMIC DNA]. Strain: K12 / W3110 / ATCC 27325 / DSM 5911. "Amino acid sequence for the peptide extension on the prolipoprotein of the Escherichia coli outer membrane." Inouye S., Wang S., Sekizawa J., Halegoua S., Inouye M. Proc. Natl. Acad. Sci. U.S.A. 74:1004-1008(1977) [PubMed: 322142] [Abstract] Cited for: PROTEIN SEQUENCE OF 1-20. "Sequence of the murein-lipoprotein and the attachment site of the lipid." Braun V., Bosch V. Eur. J. Biochem. 28:51-69(1972) [PubMed: 4261992] [Abstract] Cited for: PROTEIN SEQUENCE OF 21-78. "Covalent binding of lipid to protein. Diglyceride and amide-linked fatty acid at the Nterminal end of the murein-lipoprotein of the Escherichia coli outer membrane." Hantke K., Braun V. Eur. J. Biochem. 34:284-296(1973) [PubMed: 4575979] [Abstract] Cited for: PROTEIN SEQUENCE OF 21-24. "Poly(A) RNA in Escherichia coli: nucleotide sequence at the junction of the lpp transcript and the polyadenylate moiety." Cao G.J., Sarkar N. Proc. Natl. Acad. Sci. U.S.A. 89:7546-7550(1992) [PubMed: 1380161] [Abstract] Cited for: NUCLEOTIDE SEQUENCE [GENOMIC DNA] OF 44-78. Strain: SK5726. "Trimeric structure and localization of the major lipoprotein in the cell surface of Escherichia coli." Choi D.-S., Yamada H., Mizuno T., Mizushima S. J. Biol. Chem. 261:8953-8957(1986) [PubMed: 3013869] [Abstract] Cited for: SUBUNIT, SUBCELLULAR LOCATION. "Alterations of the carboxyl-terminal amino acid residues of Escherichia coli lipoprotein affect the formation of murein-bound lipoprotein." Zhang W.-Y., Wu H.C. J. Biol. Chem. 267:19560-19564(1992) [PubMed: 1527073] [Abstract] Cited for: MUTAGENESIS OF ASP-70; LYS-75; TYR-76; ARG-77 AND LYS-78. Strain: K12. "Lethality of the covalent linkage between mislocalized major outer membrane lipoprotein and the peptidoglycan of Escherichia coli." Yakushi T., Tajima T., Matsuyama S., Tokuda H. J. Bacteriol. 179:2857-2862(1997) [PubMed: 9139900] [Abstract] Cited for: MUTAGENESIS OF SER-22. Strain: ATCC 35607 / JM83. "TolB protein of Escherichia coli K-12 interacts with the outer membrane peptidoglycanassociated proteins Pal, Lpp and OmpA." Clavel T., Germon P., Vianney A., Portalier R., Lazzaroni J.-C. Mol. Microbiol. 29:359-367(1998) [PubMed: 9701827] [Abstract] Cited for: INTERACTION WITH TOLB. "Pal lipoprotein of Escherichia coli plays a major role in outer membrane integrity." Cascales E., Bernadac A., Gavioli M., Lazzaroni J.-C., Lloubes R. J. Bacteriol. 184:754-759(2002) [PubMed: 11790745] [Abstract] Cited for: INTERACTION WITH PAL. "TonB interacts with nonreceptor proteins in the outer membrane of Escherichia coli." Higgs P.I., Letain T.E., Merriam K.K., Burke N.S., Park H., Kang C., Postle K. J. Bacteriol. 184:1640-1648(2002) [PubMed: 11872715] [Abstract] Cited for: INTERACTION WITH TONB. Strain: K12 / W3110 / ATCC 27325 / DSM 5911. "Protein complexes of the Escherichia coli cell envelope." Stenberg F., Chovanec P., Maslen S.L., Robinson C.V., Ilag L., von Heijne G., Daley D.O. J. Biol. Chem. 280:34409-34419(2005) [PubMed: 16079137] [Abstract] Cited for: SUBUNIT, SUBCELLULAR LOCATION. Strain: BL21-DE3. "The double helix coiled coil structure of murein lipoprotein from Escherichia coli." McLachlan A.D. J. Mol. Biol. 121:493-506(1978) [PubMed: 353292] [Abstract] Cited for: 3D-STRUCTURE MODELING. "Core structure of the outer membrane lipoprotein from Escherichia coli at 1.9 A resolution." Shu W., Liu J., Ji H., Lu M. J. Mol. Biol. 299:1101-1112(2000) [PubMed: 10843861] [Abstract] Cited for: X-RAY CRYSTALLOGRAPHY (1.9 ANGSTROMS) OF 22-77, SUBUNIT. "An alanine-zipper structure determined by long range intermolecular interactions." Liu J., Lu M. J. Biol. Chem. 277:48708-48713(2002) [PubMed: 12368282] [Abstract] Cited for: X-RAY CRYSTALLOGRAPHY (1.3 ANGSTROMS) OF 22-71 OF MUTANT ALA14. "Core side-chain packing and backbone conformation in Lpp-56 coiled-coil mutants." Liu J., Cao W., Lu M. J. Mol. Biol. 318:877-888(2002) [PubMed: 12054830] [Abstract] Cited for: X-RAY CRYSTALLOGRAPHY (1.65 ANGSTROMS) OF 22-77 OF MUTANTS ALA-5 AND ALA-7. "Zinc-mediated helix capping in a triple-helical protein." Liu J., Dai J., Lu M. Biochemistry 42:5657-5664(2003) [PubMed: 12741822] [Abstract] Cited for: X-RAY CRYSTALLOGRAPHY (1.7 ANGSTROMS) OF 22-77 OF MUTANT ALA10(56). "Atomic structure of a tryptophan-zipper pentamer." Liu J., Yong W., Deng Y., Kallenbach N.R., Lu M. Proc. Natl. Acad. Sci. U.S.A. 101:16156-16161(2004) [PubMed: 15520380] [Abstract] Cited for: X-RAY CRYSTALLOGRAPHY (1.45 ANGSTROMS) OF 22-73 OF MUTANT TRP-14. Аннотация статьи "DNA sequence of the gene for the outer membrane lipoprotein of E. coli: an extremely AT-rich promoter": The outer membrane lipoprotein is the most abundant protein in an E. coli cell. Its structural gene (lpp) was cloned into a lambda phage vector and the nucleotide sequence of a DNA fragment of 814 bp encompassing the lpp gene was determined. The promoter region of the gene was found to have the following features. First, a segment of 261 bp preceding the transcription initiation site (-1 to -261) has a very high AT content of 70%, in contrast to 53% for the mRNA region of 322 bp, 44% for a segment of 127 bp after the transcription termination site and 49% for the average AT content of the E. coli chromosome. Second, in particular, of the first 45 bp upstream from the transcription initiation site (-1 to -45), 36 bases (80%) are A or T. Third, there is a heptanucleotide sequence homologous to the "Pribnow box," eight bases apart from the transcription initiation site. Fourth, a sequence homologous to the "RNA polymerase recognition site" exists on both strands between positions -27 and -39. Finally, there is a long dyad symmetry centered at the transcription initiation site. Перевод аннотации: Последовательность гена липопротеина наружной мембраны E. coli: очень ATбогатый промотор Липопротеин наружной мембраны - самый частый белок в клетке E. coli. Его структурный ген (lpp) был клонирован в вектор фага лямбды, и нуклеотидная последовательность фрагмента ДНК протяженностью 814 пар нуклеотидов, охватывающая ген lpp, была определена. Было обнаружено, что промоторная область гена обладала следующими особенностями. 1. Участок протяженностью 261 пара нуклеотидов, предшествующий сайту инициации транскрипции (с -1 до -261), имеет очень высокое содержание AT (70%). Для сравнения: область мРНК протяженностью 322 пары нуклеотидов содержит 53% AT, посттерминационная область протяженностью 127 пар нуклеотидов содержит 44% AT. В среднем, хромосома E. coli содержит 49% AT. 2. Из 45 пар нуклеотидов к N-концу от сайта инициации (с -1 до -45), 36 пар нуклеотидов (80%) являются A или T. 3. Есть шестинуклеотидная последовательность, гомологичная "Pribnow box" (8 пар оснований, стоящие в стороне от сайта инициации трансляции). 4. Последовательность, гомологичная "сайту узнавания РНК-полимеразой", расположена на обеих нитях ДНК (прямой и обратной) между позициями -27 и -39. Наконец, есть протяженная симметрия пар с центром в сайте инициации транскрипции.